应用|乙肝新药开发应用,基于抑制cccDNA形成,RNAi理论基础
在介绍众多科研力量投入慢性乙肝新药开发之际 , 不得不提的既是RNAi这项前沿技术 。 RNA干扰是如何在治疗慢性乙肝研究药物中脱颖而出的呢?我们先来了解一下 , 乙肝病毒是如何复制的 。

文章图片
乙肝新药开发应用 , 基于抑制cccDNA形成 , RNAi理论基础
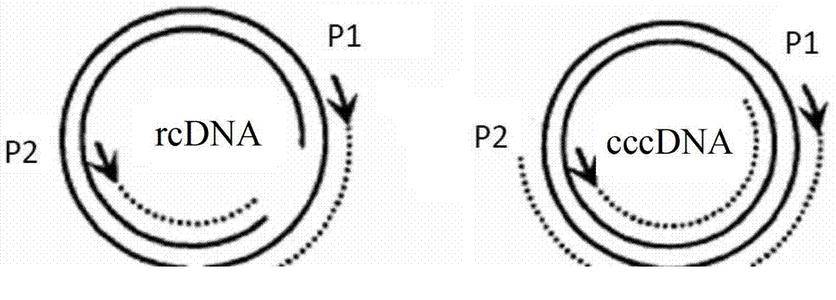
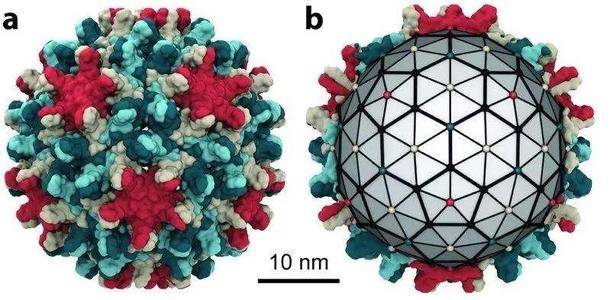
乙肝病毒(HBV)的病毒粒子 , 或Dane颗粒 , 有大、中、小表面抗原嵌入周围的包膜 。 核衣壳位于被膜内 , 由一个20面体衣壳组成 , 该衣壳通常由120个核心蛋白二聚体组成 , 带有包裹的病毒松弛环状DNA(rcDNA)和聚合酶蛋白 。 除了完整的感染颗粒外 , 乙肝病毒感染者的血清中还含有大量的亚病毒颗粒 , 这些亚病毒颗粒主要由小的表面蛋白组成 。
【应用|乙肝新药开发应用,基于抑制cccDNA形成,RNAi理论基础】肝细胞感染由Dane颗粒与位于细胞表面的糖胺聚糖相互作用引起的 。 此后 , 肉豆蔻大表面抗原与牛磺胆酸钠(NTCP)胆汁酸转运体的特异性结合启动了核衣壳的细胞进入 。 已有证据表明 , 表皮生长因子受体也可能在乙肝病毒进入肝细胞的吸收中发挥作用 。 HBV的NTCP受体直到2012年才被发现 , 并为HBV复制研究提供了宝贵的动力 。
文章图片
在这一发现之前 , 全球研究人员由于缺乏关于乙肝病毒如何在感染过程中 , 进入肝细胞的信息而阻碍了对乙肝病毒的研究 。 在上一章节 , 小番健康已经初步介绍了RNA干扰(RNAi)激活剂对HBV生命周期的破坏机制 , 更深层的讲 , 衣壳被调控分解后释放出rcDNA , 然后在细胞核内转化为共价闭合环状DNA(cccDNA) 。 修复过程包括使用宿主细胞酶 , 如酪氨酸DNA磷酸二酯酶2(TDP2)和皮瓣内切酶1(Fen1) 。
将rcDNA转化为cccDNA和建立HBV复制 , 是一个有效的过程 。 南非约翰内斯堡威特沃特斯兰德大学健康科学学院病理学院抗病毒基因治疗研究室研究人员介绍 , 在对黑猩猩和鸭子的动物模型研究表明 , 一个单一的感染粒子就足以引发复制病毒的感染 。 cccDNA是一种稳定的复制中间产物 , 作为编码mRNAs的前基因组RNA(pgRNA)和病毒蛋白的转录模板 。
文章图片
维持感染肝细胞内cccDNA , 是由HBx由X开放阅读框(ORF)编码的蛋白质) 。 染色体外DNA(如cccDNA)被5/6染色体(SMC5/6)复合体的细胞结构维持作为转录沉默的靶点 。 HBx与泛素连接酶1的受损DNA结合域相互作用 , 使SMC5/6不稳定 , 从而促进了HBV基因表达 。 cccDNA的orf排列在一个非常紧凑的组织中 。 蛋白质编码序列相互重叠 , 多蛋白编码通过使用不同的读取帧来实现 。
进一步增加紧凑的安排是嵌入调控元素 , 比如启动子和增强子 , 在病毒性开放阅读框 。 了解完整的cccDNA后 , 我们再来了解基于RNAi抗HBV治疗的理论基础 。 RNA分子 , 包括编码病毒蛋白的pgRNA和mRNAs , 对HBV复制至关重要 。 因此 , 破坏这些病毒序列的稳定性 , 被认为是灭活病毒的好策略 。 这种治疗方法通过降解pgRNA , 抑制cccDNA的形成 。
此外 , 抑制对cccDNA形成很重要的病毒蛋白的翻译 , 比如C和HBx , 也会限制cccDNA的产生 。 HBV基因组的紧凑排列限制了序列的可塑性和逃避沉默序列的能力 。 此外 , 包含HBx的常见3′序列的存在 , 意味着不同的乙肝病毒转录物可能被个体沉默序列所靶向 , 这就是当前全球研究人员推进基于RNAi开发慢性乙肝新药的理论基础 。
- 新药|乙肝在研新药ABI-H0731,II期211研究,安全性数据
- 强而|乙肝RNAi新药机制,强而特异性基因沉默,受小双链RNA影响
- 小布|集纳700万条电子病历数据,复旦儿科“AI医生”进入临床应用
- 澎湃新闻|《新药的故事2》:理性看待疫情 也要保持警觉
- 算法|谷歌糖尿病视网膜病变AI检测算法,已在印度和泰国医院应用
- 研究|乙肝在研新药2种化合物,小鼠模型,组合或联合抗HBV药物价值
- 药物|乙肝重新认识,NA抑制新cccDNA形成,1种新药进入3期
- 古巴|乙肝在研新药NASVAC,日本古巴科学家开发,鼻腔给药改良版
- 测量|乙肝现实目标,彻底清除或需联药,测量肝内cccDNA是难点
- 英特尔|英特尔与京东优化分布式特征向量检索系统Vearch,加速AI应用创新
